-
Notifications
You must be signed in to change notification settings - Fork 27
Commit
This commit does not belong to any branch on this repository, and may belong to a fork outside of the repository.
- Loading branch information
Showing
1 changed file
with
155 additions
and
0 deletions.
There are no files selected for viewing
This file contains bidirectional Unicode text that may be interpreted or compiled differently than what appears below. To review, open the file in an editor that reveals hidden Unicode characters.
Learn more about bidirectional Unicode characters
| Original file line number | Diff line number | Diff line change |
|---|---|---|
| @@ -0,0 +1,155 @@ | ||
| --- | ||
| date: 2024-08-18 | ||
| comments: true | ||
| --- | ||
| # 生信爱好者周刊(第 136 期):从错误中学习 | ||
|
|
||
| 这里记录每周值得分享的生信相关内容,周日发布。 | ||
|
|
||
| 本杂志开源(GitHub: [openbiox/weekly](https://github.com/openbiox/weekly "openbiox/weekly")),欢迎提交 issue,投稿或推荐生信相关内容。 | ||
|
|
||
| [「生信周刊讨论区」](https://github.com/openbiox/weekly/discussions "「生信周刊讨论区」") | ||
|
|
||
| ## 封面图 | ||
|  | ||
| 从左到右分别为“从理论学习”、“从实践学习”、“从错误学习”。 | ||
|
|
||
| ## 本周话题:从错误中学习 | ||
|
|
||
| @科技爱好者周刊 如本期的封面图,从左到右分别为“从理论学习”、“从实践学习”、“从错误学习”。这张图的意思是,书本知识只是基础,实践应用可以学到更多,如果实践发生错误,那就是最好的学习机会。 | ||
|
|
||
| @kkjtmac 从错误中学习是成长的重要途径,通过错误能获得深刻的经验和教训。然而,单靠犯错并不可取,理论和实践也是必不可少的基础。我们应在日常工作中多总结错误,结合理论和实践,提升整体学习效果。 | ||
|
|
||
| ## 生信研究 | ||
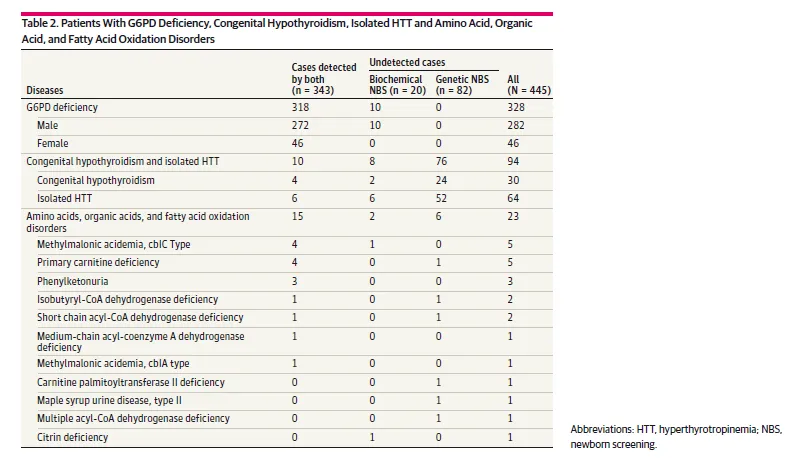
| 1、[JAMA Network | 近3万例新生儿!大规模前瞻性研究表明:基因测序或可作为新生儿一级筛查的关键方法](https://mp.weixin.qq.com/s/SZMqjkoFBwhg7KDQyFPnsw) | ||
|
|
||
|  | ||
|
|
||
| 《JAMA Network Open》这篇题为《Genomic Sequencing as a First-Tier Screening Test and Outcomes of Newborn Screening》的文章为基因筛查作为一级筛查方法来改进现有新生儿筛查项目提供了重要的数据支撑,对临床医师、遗传学家和家庭提供更好的医疗保健和咨询服务具有重要意义。据了解,这是目前最大规模的评估基因测序用于新生儿一级筛查潜在益处的前瞻性研究。 | ||
|
|
||
| - 论文链接:https://jamanetwork.com/journals/jamanetworkopen/fullarticle/2809067 | ||
|
|
||
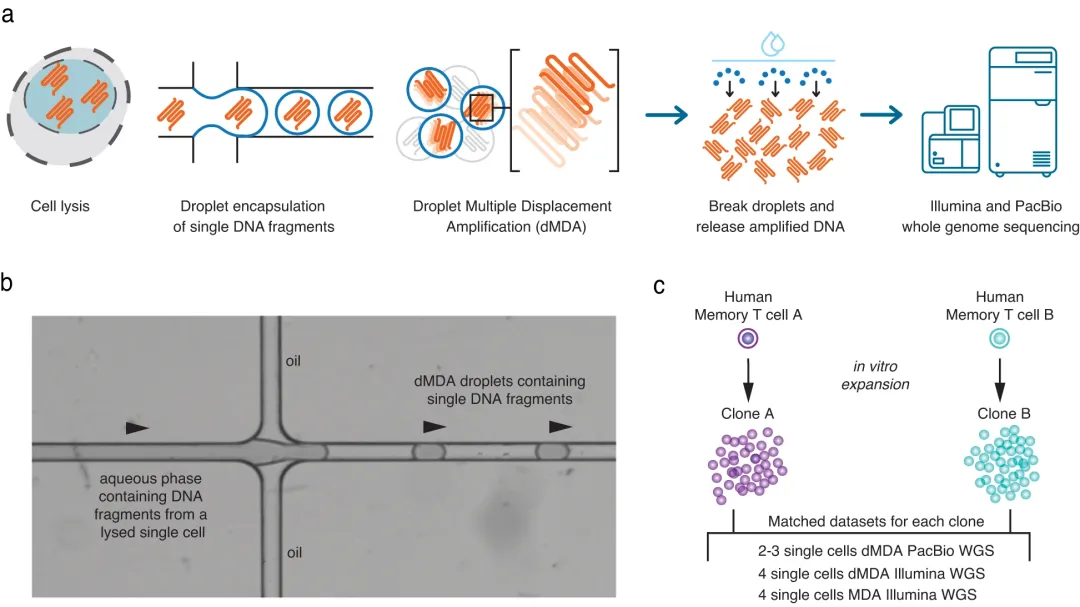
| 2、[Nature Communication | 人类单细胞长读长全基因组分析新方法,可为单细胞遗传变异提供新见解](https://mp.weixin.qq.com/s/ofm3ETTWbDzjshbFvRYv1w) | ||
|
|
||
|  | ||
|
|
||
| 在过去几年中,长读长测序技术在通量和数据质量方面取得了显著进步,极大地促进了人们对人类基因组变异的理解。长读长测序能够读取重复和高GC含量的区域,对于生成真核生物多样性的参考基因组,以及绘制完整人类基因组端粒到端粒图谱至关重要。长读长测序的另一个优点是能够对难以或无法通过其他基因组测序方法识别的复杂结构变异(SV)和重复元件进行基因分型。 | ||
|
|
||
| 人类单细胞全基因组测序(WGS)已成为一个热点研究领域,有助于解答细胞生物学多个领域的基本问题。目前,单细胞WGS研究主要集中在表征短读长测序中可检测到的遗传变异,包括单核苷酸变异(SNV)等。虽然单细胞短读长全基因组测序有多种方法,但仍未有识别单细胞全部遗传变异的技术。 | ||
|
|
||
| 2023年8月,瑞典乌普萨拉大学等多个机构的研究人员在Nature Communications发表了题为“Long-read whole-genome analysis of human single cells”的文章,提出了一种新的人类单细胞长读长全基因组分析方法。利用基于PacBio高保真(HiFi)测序平台的长读长测序,对人类单个CD8+ T细胞进行WGS分析,并通过多重置换扩增(MDA)获得足以进行长读长测序的DNA。结果表明,与短读长测序相比,长读长测序可提高单细胞遗传变异检测的性能,特别是检测单倍型、复杂结构变异和串联重复序列(TR);并证明从头重建部分人类T细胞基因组是可行的。 | ||
|
|
||
| - 论文链接:https://www.nature.com/articles/s41467-023-40898-3 | ||
|
|
||
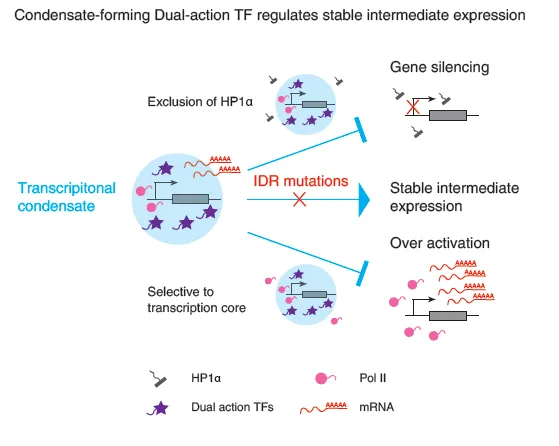
| 3、[Cell | 李寅青/李丕龙团队合作揭示基因表达的“保温杯”——开发全基因组范围内检测相分离分布的技术](https://mp.weixin.qq.com/s/L5yEJrCdo23TAMHyAcinnQ) | ||
|
|
||
|  | ||
|
|
||
| 2024年4月16日,清华大学药学院李寅青课题组与清华大学生命科学学院李丕龙课题组在Cell杂志上发表题为Dual-Role Transcription Factors Stabilize Intermediate Expression Levels的文章。该研究开发了一种名为Assay for Chromatin-bound Condensates by exploratory Sequencing(ACC-Seq)的全基因组范围内检测凝聚体分布的技术。通过ACC-Seq,研究人员鉴定了一类独特的双效应转录因子(dual-action TFs)。这类转录因子通过形成转录凝聚体,能在抑制基因高表达的同时激活沉默状态的基因,从而将基因表达水平“保温”在特定的活化程度。 | ||
|
|
||
| - 论文链接:https://doi.org/10.1016/j.cell.2024.03.023 | ||
|
|
||
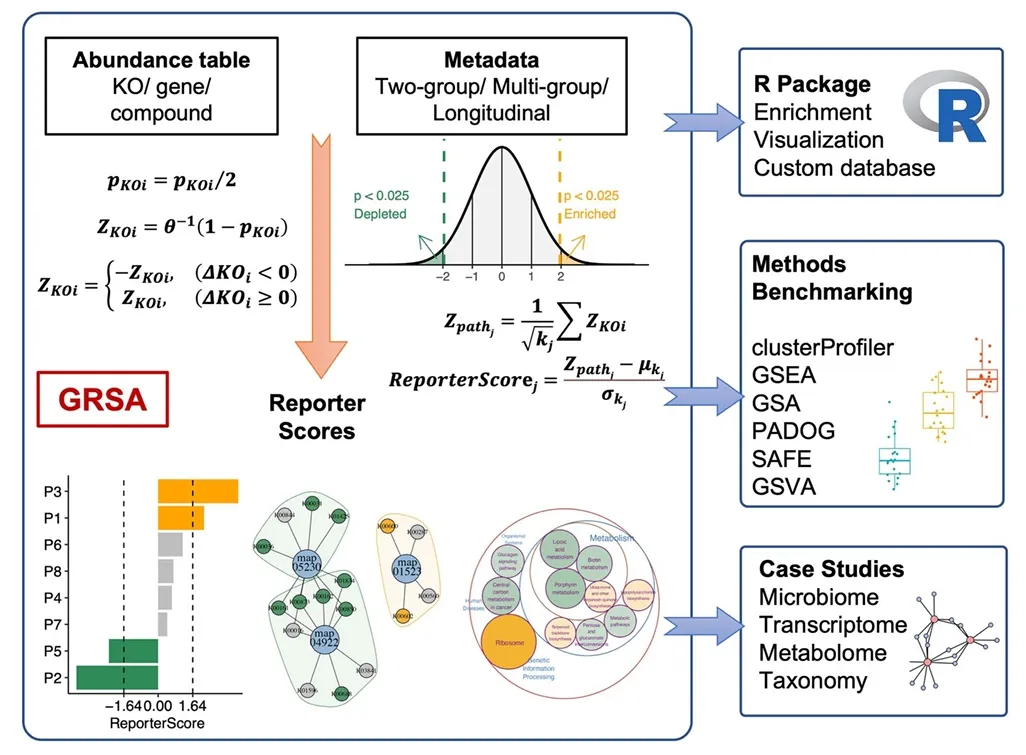
| 4、[Briefings in Bioinformatics | 面向多组及纵向实验设计的各类组学数据的功能富集新方法 GRSA](https://mp.weixin.qq.com/s/EMY0r3DV_orO1FUl1BcLlQ) | ||
|
|
||
|  | ||
|
|
||
| 富集分析将生物特征置于通路中,促进对高维数据的系统理解,在生物医学研究中被广泛应用。然而,大多数现有工具无法直接将富集分析应用于多组和纵向组学数据。新兴的基于报告评分富集分析(RSA)方法显示出卓越的敏感性,因为它依赖于p值而不是特征原始值,但由于缺乏适当的工具,它经常被误用。我们提出了面向多组和纵向组学数据的广义报告评分分析(Generalized Reporter Score-based Analysis,GRSA)方法。与其他常用的富集分析方法进行比较表明,GRSA在多个基准数据集上具有更高的敏感性。我们将GRSA应用于微生物组、转录组和代谢组数据,并在组学研究中发现了新的生物学见解。最后,我们使用 GRSA 在分类数据库进行富集,展示其应用不仅限于功能富集。我们开发了一个R包:ReporterScore,在其中实现GRSA,并与强大的可视化模块和可更新的通路数据库集成在一起(https://github.com/Asa12138/ReporterScore)。我们相信GRSA方法和ReporterScore包将成为生物医学研究领域中的宝贵资源。 | ||
|
|
||
| - 论文链接:https://doi.org/10.1093/bib/bbae116 | ||
|
|
||
| ## 博文资讯 | ||
| 5、[从四个方向理解AI](https://mp.weixin.qq.com/s/I1RMxTTtQ4UbiJC_r5_R4Q) | ||
|
|
||
|  | ||
|
|
||
| 2024年3月29日,中信出版集团联合上海交通大学上海高级金融学院邀请到凯文·凯利(K.K.)来到中国,展开一场精彩的线下对谈。读者们熟悉的K.K.,是《连线》杂志的创始主编,是《失控》《必然》《5000天后的世界》等畅销书的作者。除了科技预言式写作,去年K.K.也正式出版了一本凝结了他 70 年人生经验的智慧箴言录——《宝贵的人生建议》。 | ||
| 现场,K.K. 以《What's Next:in AI and Digital World》为主题带来了他对于全球科技下一步发展趋势的深入思考和精彩见地,讨论了关于谁是AI的第一批使用者、AI与人类所产生的情感羁绊等问题。 | ||
|
|
||
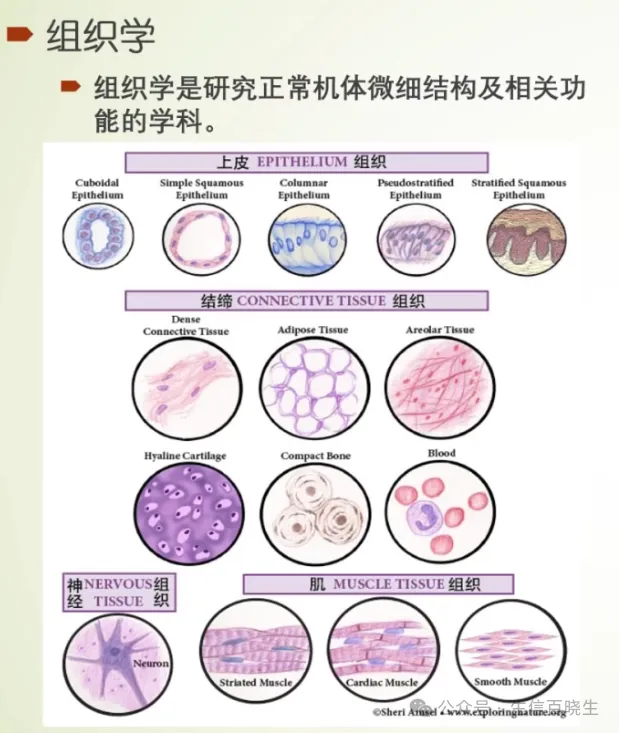
| 6、[单细胞注释前的课前准备 --组织类型与细胞类型](https://mp.weixin.qq.com/s/GR55MNRNKhbcQiWZJJ9bvA) | ||
|
|
||
|  | ||
|
|
||
| 单细胞注释前,了解组织的特点和可能存在的细胞类型、相对比例,对细胞注释会有很大的帮助。 | ||
| 本文整理主要参考:贺晓舟老师的 人体组织学 | ||
|
|
||
| - 视频链接:https://www.bilibili.com/video/BV1B841187EE/?spm_id_from=333.788&vd_source=5192954f066222a36e500187eb9825f3 | ||
|
|
||
| 7、[生命是怎样涌现的:系统生物学入门全路径](https://mp.weixin.qq.com/s/IHrWEmZ3mh-KXChHaaHy-w) | ||
|
|
||
|  | ||
|
|
||
| 系统生物学(Systems biology),是一个使用整体论(而非还原论)研究范式,整合不同学科、层次的信息以理解生物系统如何行使功能的学术领域,是分子生物学之后现代生物学的全新阶段,包括表观遗传学、各种生物组学、合成生物学、生物信息学等细分领域。 | ||
|
|
||
| ## 工具 | ||
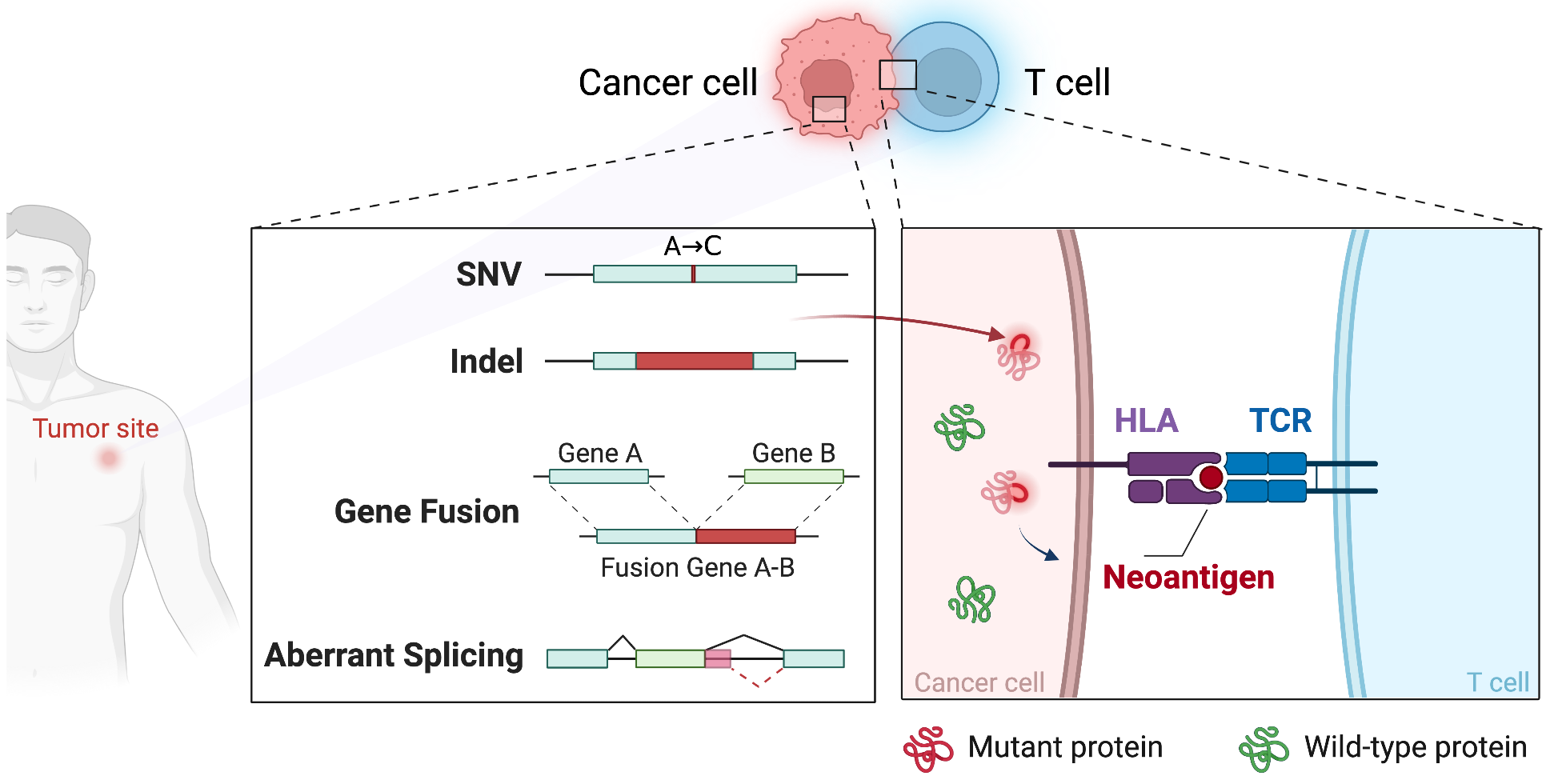
| 8、[NeoHunter | 肿瘤新抗原检测软件](https://mp.weixin.qq.com/s/QaTk2kv5KIqE0e5R2lYcFQ) | ||
|
|
||
|  | ||
|
|
||
| NeoHunter是用于全面检测肿瘤新抗原的软件,能从测序数据中检测多种肿瘤新抗原,包括SNV、插入/缺失、基因融合等,支持个性化疫苗开发。 | ||
| - 论文链接:https://onlinelibrary.wiley.com/doi/full/10.1002/qub2.28 | ||
| - 工具链接:https://github.com/XuegongLab/NeoHunter | ||
|
|
||
| 9、[LightMirrors | 开源的缓存镜像站服务](https://github.com/NoCLin/LightMirrors "LightMirrors | 开源的缓存镜像站服务") | ||
|
|
||
|  | ||
|
|
||
| LightMirrors是一个开源的缓存镜像站服务,用于加速软件包下载和镜像拉取。 目前支持 DockerHub、 K8s、 GitHub Container Registry、 Quay.io、 PyPI、 PyTorch、 NPM、 GoProxy 等镜像缓存服务。 | ||
|
|
||
| - 工具链接:https://github.com/NoCLin/LightMirrors | ||
|
|
||
|
|
||
| 10、[AGAT | 处理GTF/GFF格式基因注释的工具包](https://github.com/NBISweden/AGAT "AGAT | 处理GTF/GFF格式基因注释的工具包") | ||
|
|
||
|  | ||
|
|
||
| AGAT(Another Gtf/Gff Analysis Toolkit)是一个Perl脚本工具集,专门用于处理GTF/GFF格式的基因注释文件。它能够标准化、清洗、转换格式、合并、修改、过滤注释,并支持从FASTA序列中提取信息等多种功能,适合处理复杂或存在错误的基因注释数据。 | ||
| - 工具链接:https://github.com/NBISweden/AGAT | ||
|
|
||
| 11、[TFTF | 转录因子-靶基因综合分析预测R包/App](https://mp.weixin.qq.com/s/gWvwI5Tx8e4IDZjpT8Fusg) | ||
|
|
||
|  | ||
|
|
||
| TF-Target Finder (TFTF) 是一个新发表的R包/App,用于预测转录因子的靶基因和靶基因的上游转录因子。它集成了多个数据库和算法,通过相关性分析,提高预测转录因子-靶基因调控关系的可靠性,并提供了可视化界面。 | ||
| - 网页链接:https://jingle.shinyapps.io/TF_Target_Finder/ | ||
| - R包链接:devtools::install_github("WangJin93/TFTF") | ||
|
|
||
| ## 资源 | ||
| 12、[chapin-books | 技术书籍和资源GitHub仓库](https://github.com/chapin666/books "chapin-books | 技术书籍和资源GitHub仓库") | ||
|
|
||
| GitHub仓库"chapin666/books"是一个收藏夹,包含了多个领域的技术书籍和资源,如算法、架构、操作系统、数据库等,旨在为开发者和技术爱好者提供丰富的学习材料。 | ||
|
|
||
| - 资源链接:https://github.com/chapin666/books | ||
|
|
||
| 13、[2024年最佳12款开源知识库软件](https://helpjuice.com/blog/open-source-knowledge-base#10-xwiki-11 "2024年最佳12款开源知识库软件") | ||
|
|
||
|  | ||
|
|
||
| 这里列举了2024年最佳12款开源知识库软件包括BookStack、OpenKM等,各有特点如易用性、自托管、高度可定制。它们支持企业构建知识管理系统,提供信息存储、搜索、分析等功能,且多为免费,适合不同规模和需求的企业使用。 | ||
|
|
||
| - 资源链接:https://helpjuice.com/blog/open-source-knowledge-base#10-xwiki-11 | ||
| ## 历史上的本周 | ||
|
|
||
| - 第96期:[如何取得杰出成就](https://mp.weixin.qq.com/s/DtrUrAycYijB4nNmTdX9zQ) | ||
|
|
||
| ## 贡献者(GitHub ID) | ||
|
|
||
| 「Openbiox 生信周刊」运维小队: | ||
|
|
||
| - `@ShixiangWang`(王诗翔) | ||
| - `@kkjtmac`(阚科佳) | ||
| - `@NiEntropy`(赵启祥) | ||
| - `@He-Kai-fly`(何凯) | ||
| - `@JnanZhang`(张佳楠) | ||
| - `@Tomcxf`(陈啸枫) | ||
| - `@wangdepin`(王德品) | ||
| - `@kongjianyang`(空间阳) | ||
|
|
||
| ## 订阅 | ||
|
|
||
| 这个周刊每周日发布,同步更新在微信公众号「优雅R」(elegant-r)上。 | ||
|
|
||
| 微信搜索“优雅R”或者扫描二维码,即可订阅。 | ||
|
|
||
|  | ||
|
|
||
| (完) |