| date | comments |
|---|---|
2023-03-16 |
true |
这里记录每周值得分享的生信相关内容,周日发布。
本杂志开源(GitHub: openbiox/weekly),欢迎提交 issue,投稿或推荐生信相关内容。
本周话题:选人不选项目的「基石项目」能否走向成功?
它面向中国内地及港澳地区全职工作的科研人员,每人每年最高500万经费,连续资助5年。5年周期一过,还有机会再续!而且它只选人,不选项目。换言之,它专注于支持富有创造力的科学家开展探索性与风险性强的基础研究。
如何选?科学家主导原则。由施一公带头,潘建伟、谢晓亮、张杰等知名科学家组成的科学委员会来决定。
@ShixiangWang - 从科学探索奖到新基石,我们看到越来越多的企业开始在基础科学研究中发力发钱,本次的新基石项目聚焦于从“0”到“1”的研究,即「项目耗时长,风险性高,本身成果就不容易产出,也就不那么容易得到资助」,实际申请和评审过程中什么程度算是“0”的开始,中国杰出青年人才中真正有多少人有做“0”到“1”研究的想法和胆量都没有数据支持,所以是否能成功目前应当持观望状态:第1-2届获选课题的想法、内容以及未来五年的进展如何将会直接影响该项目的成功度。会不会胎死腹中,或是逐步走出完整的中国商业对基础研究的支持体系,值得期待。
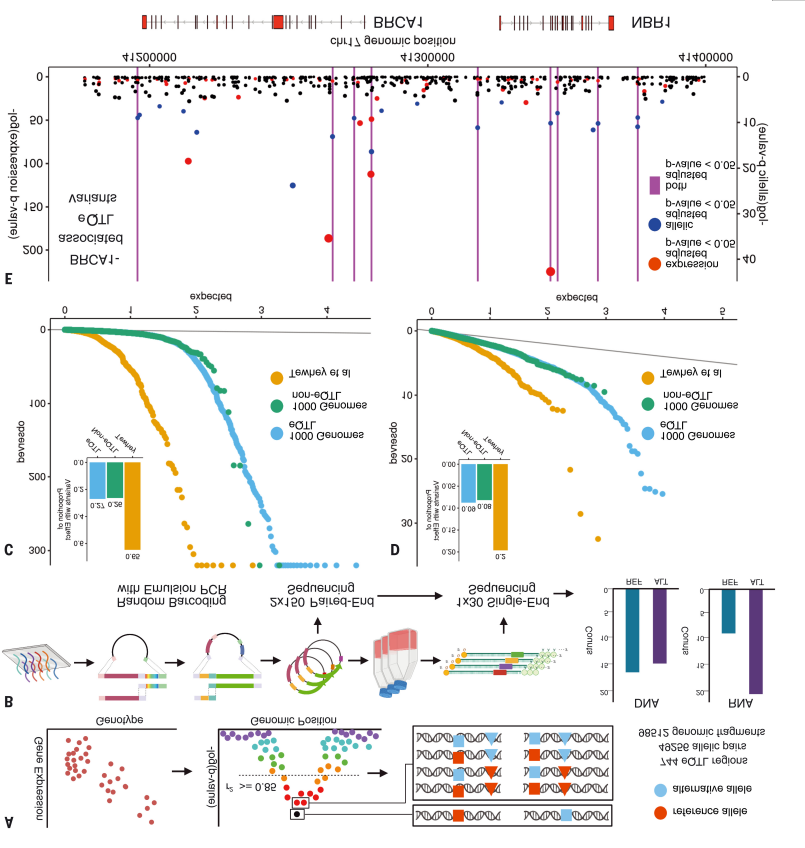
1、Science | 大规模平行报告分析(MPRA)确定多种因果变异与人类表型的关联
坦福大学医学院研究团队证明,MPRAs提供了一个可扩展的平台来分离和绘制复杂表型相关自然遗传变异的调控事件,并强调了现有方法的局限性。研究发现,显著和非显著等位基因MPRA变异相对于相应的全基因组分布呈现高度移动的特点。这一效应表明,功能预测可能能够很容易地从基因组背景中区分eQTL区域,同时区分同一区域内高度关联和共同显著变异的调控。以上结果为包括基因表达和复杂表型在内的数百种独立遗传相关特定变异研究提供了新的证据。
论文链接:https://www.science.org/doi/10.1126/science.abj5117
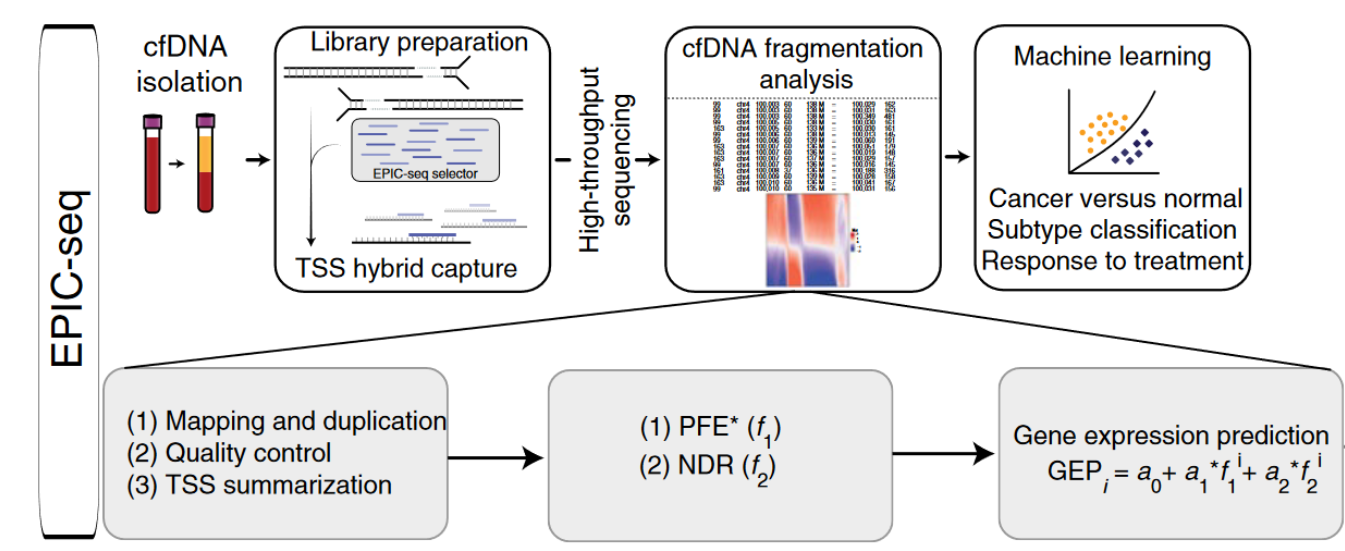
2、Nature Biotechnology | cfDNA片段化模式新方法EPIC-seq实现癌症高灵敏度检测和亚型区分
cfDNA是释放到血浆中的降解DNA片段,能够随着组织损伤、癌症和炎症反应等发生浓度变化,在疾病的早期诊断、预后、监测等方面具有重要潜在价值。
发表在Nature Biotechnology上的一篇研究介绍了一种基于cfDNA检测癌症的新方法——EPIC-seq。通过捕获目标基因的转录起始位点TSS区域,实现靶向深度测序;机器学习结合cfDNA分子特征——启动子碎片熵(PFE)和核小体缺失区 (NDR)深度,构建预测模型,预测目标RNA表达。在非小细胞肺癌(NSCLC)和弥漫性大B细胞淋巴瘤中,通过该方法实现了高灵敏度检测和亚型区分。
论文链接:https://www.nature.com/articles/s41587-022-01222-4
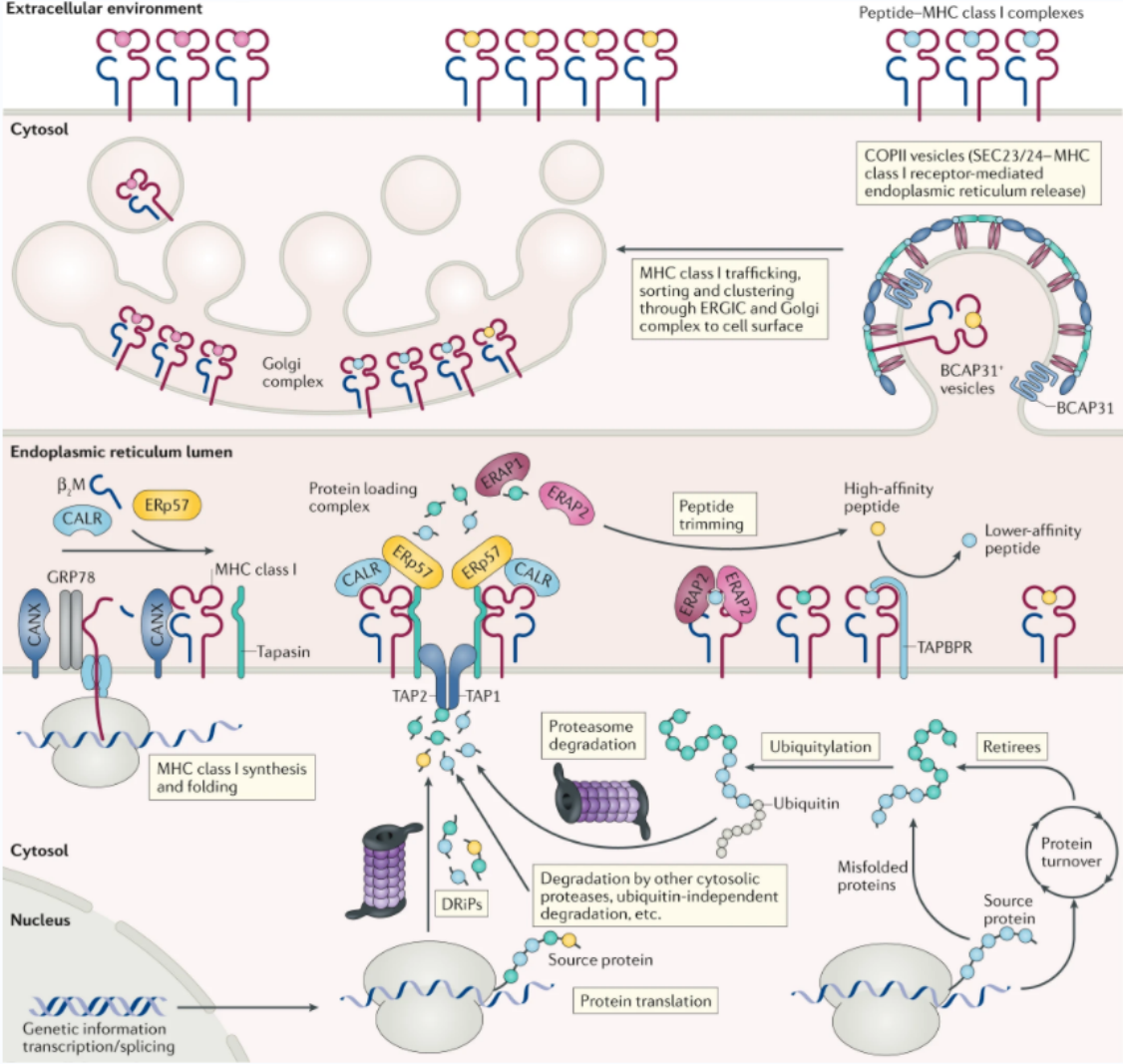
3、Nature Review Immunol. | MHCⅠ类分子与肿瘤免疫监视和免疫逃避
在这篇综述中,作者回顾了限制CD8+T细胞免疫疗法应用的两个因素:①难以识别MHC I类分子提呈的肿瘤特异性肽;②肿瘤细胞在T细胞选择下进化时损害抗原提呈的能力。作者详细描述了在有缺陷的核糖体产物的非规范翻译如何产生肽方面的最新进展,将其与作为致癌特征的翻译失调相关联,并提出将失调翻译作为肿瘤特异性肽的重要新来源。另外,作者还讨论了抗原加工和提呈途径的成分(包括最近描述的免疫核糖体)的合成和功能如何被肿瘤操纵以进行免疫逃逸,并指出可能增强免疫治疗的常见药物靶点。
论文链接:https://www.nature.com/articles/s41577-020-0390-6
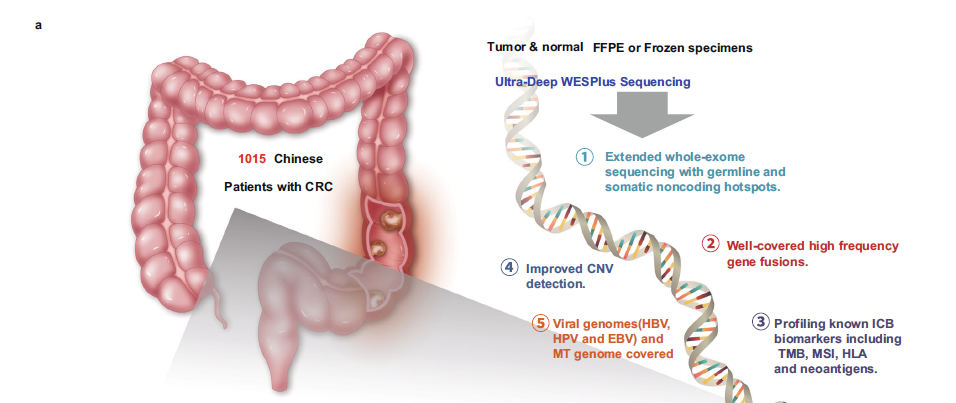
4、Nature Communication | 我国千人肠癌基因组新分型
中山大学肿瘤防治中心徐瑞华/王峰团队在Nature Communications发表的研究,全面分析了1015例中国结直肠癌患者的染色体基因组变异事件、免疫原性特征以及线粒体基因组特点,并联合临床信息探究结直肠癌分子特征和临床特征的关系。 测序原始数据已上传至中国国家生物信息中心国家基因组数据中心(HRA000873),临床信息在Supplementary Data2(OS只给了生存状态,没发现生存时间),数据库中提供了处理好的maf文件。
数据库链接:https://changkang.hapyun.com/
论文链接:https://www.nature.com/articles/s41467-022-30062-8
5、iMeta | Majorbio Cloud:一站式多组学数据分析平台
Majorbio Cloud是一个高通量多组学分析的在线平台,自带的分析流程涵盖了微生态、转录组、蛋白与代谢及基因组等多组学。通过详尽的在线文档和在线课堂,使用者可以更加简单快速的上手使用。此外,该平台也为用户提供了丰富的可视化插件库。目前Majorbio Cloud已有1200+文章引用了。
论文链接:https://onlinelibrary.wiley.com/doi/10.1002/imt2.12
话说这个imeta期刊最近老是喜欢推一些商业公司生信云平台弄成的文章。
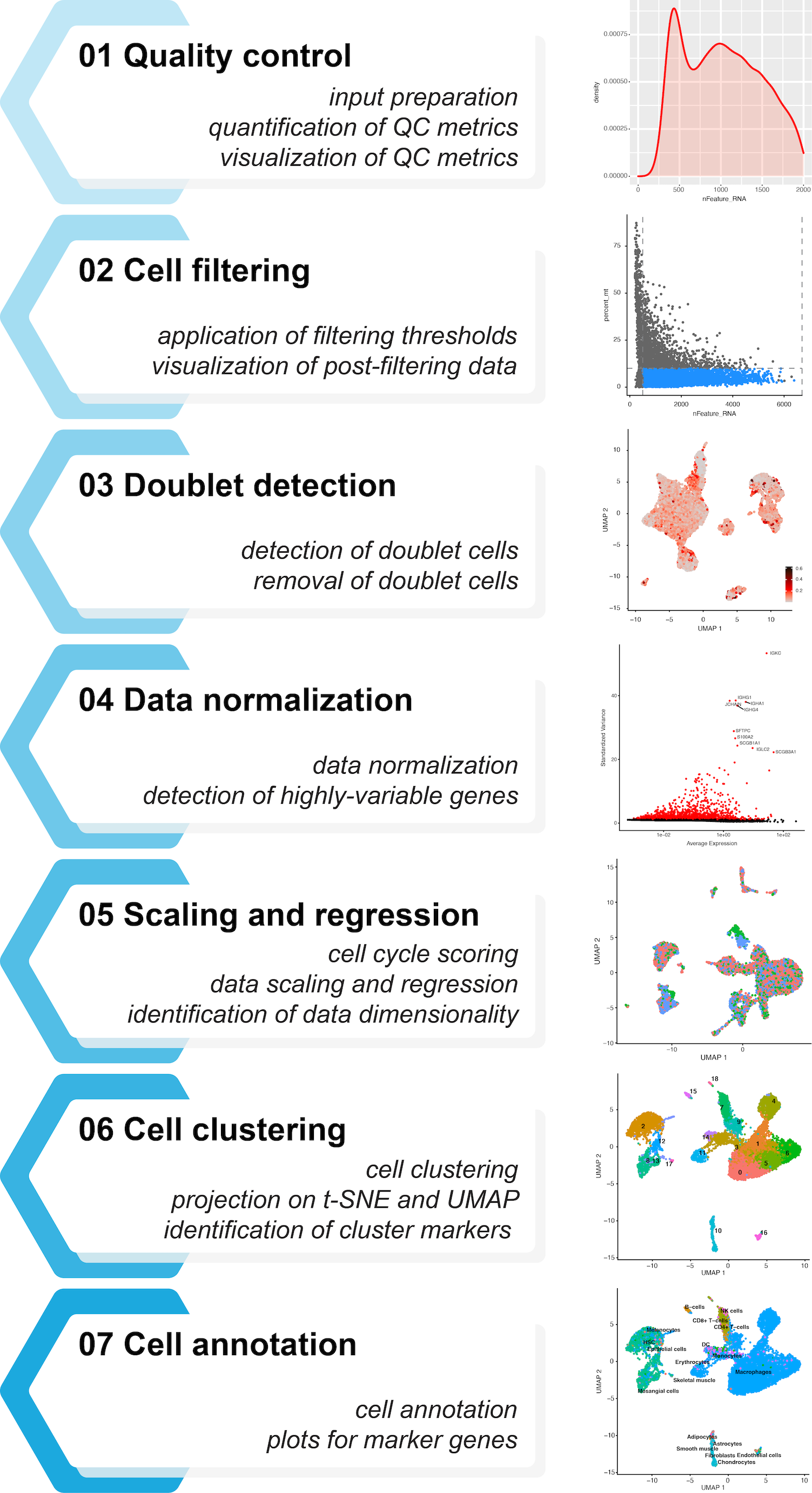
本文通过示例数据,从单细胞的质控到最后的细胞注释,展示了如何使用popsicleR包进行单细胞转录组一条龙分析
Github: https://github.com/bicciatolab/popsicleR
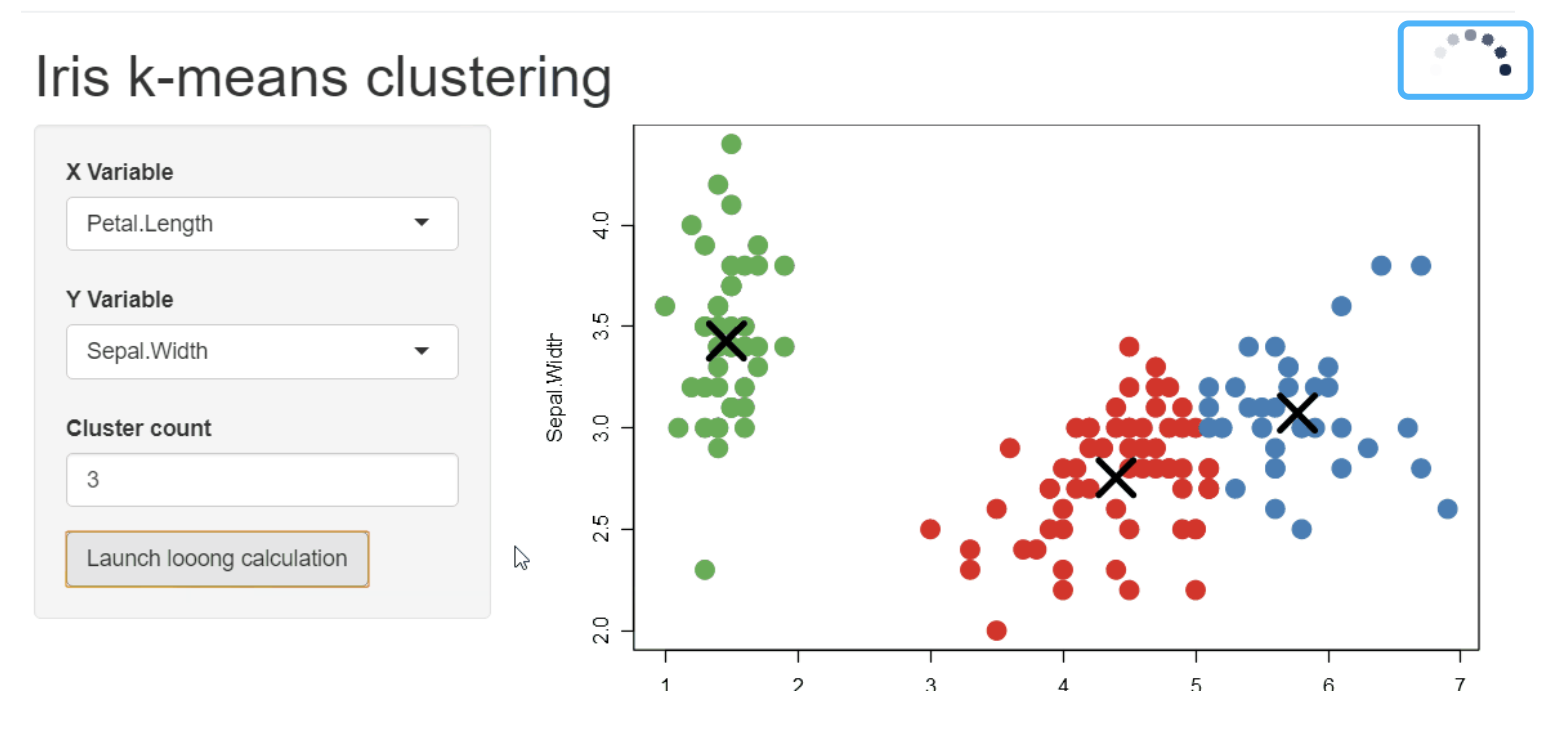
7、推荐 10 个好用到爆的 Jupyter Notebook 插件
Jupter Notebook 是基于网页的用于交互计算的应用程序,可以在网页中直接编写和运行代码,具有灵活、高度可扩展的特性,用户可通过安装插件轻松实现一些实用的功能,而本文介绍几个在jupyter notebook中被广泛用到的插件,能够更好地帮助大家进行数据分析可视化,大大提高日常的工作效率!!
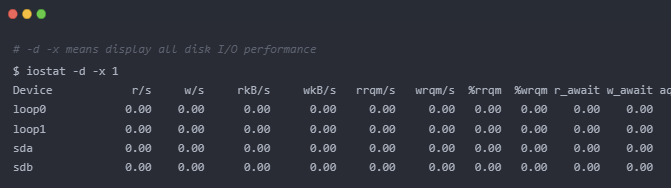
本文介绍了Linux的磁盘性能指标,并介绍了获取磁盘 I/O 情况的命令iostat,以及观察进程的 I/O 情况的命令pidstat、iotop。
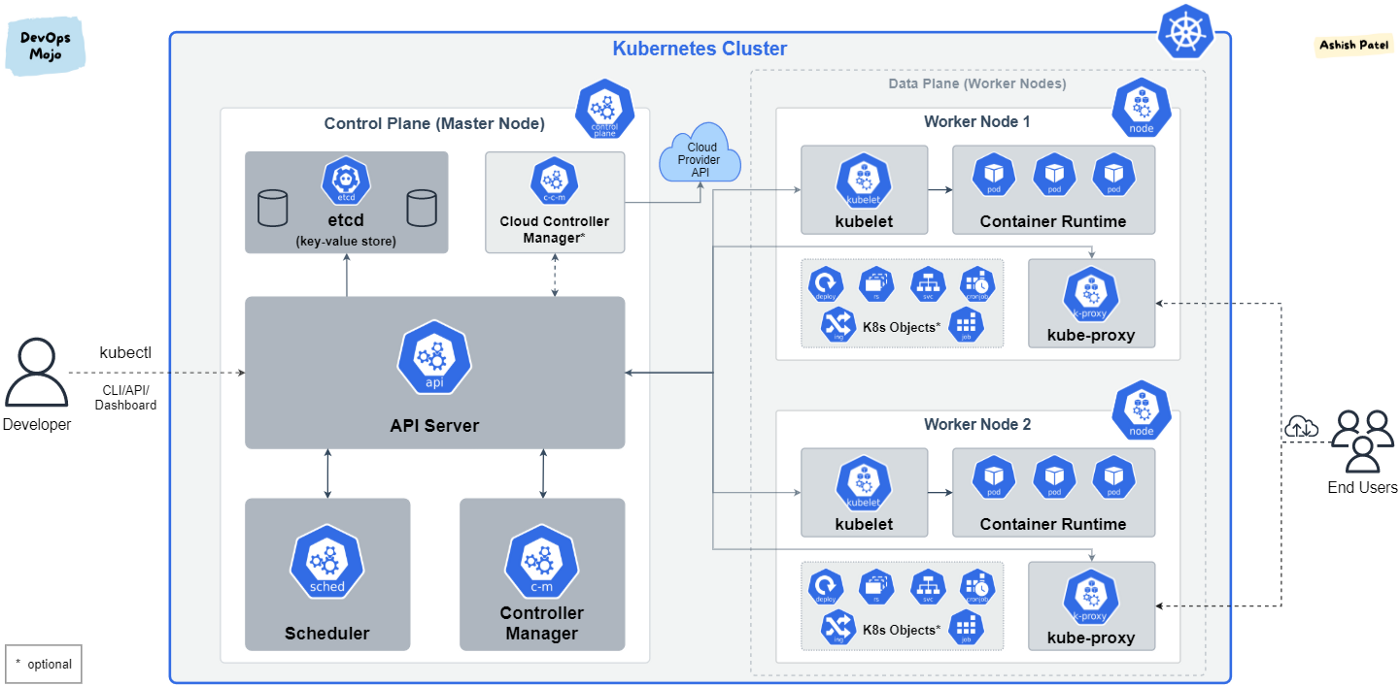
通过本推文,你可以了解到什么是微服务、容器和Kubernetes,以及他们之间的联系与区别。
10、https://github.com/hpc/Spindle
Spindle是一款用于提高HPC环境中动态库和python加载的性能的工具,可以对集群中的上千个进程进行有效管理。
Simpr包提供了一个通用、简单清晰且友好的框架,用于生成模拟数据,拟合模型和整理模型结果,完整的工作流可以在单个pipeline中完成,且不需外部函数的创建以及全局变量和循环的使用,该包功能对一些功率分析、设计分析、仿真研究和教学统计数据十分有用~
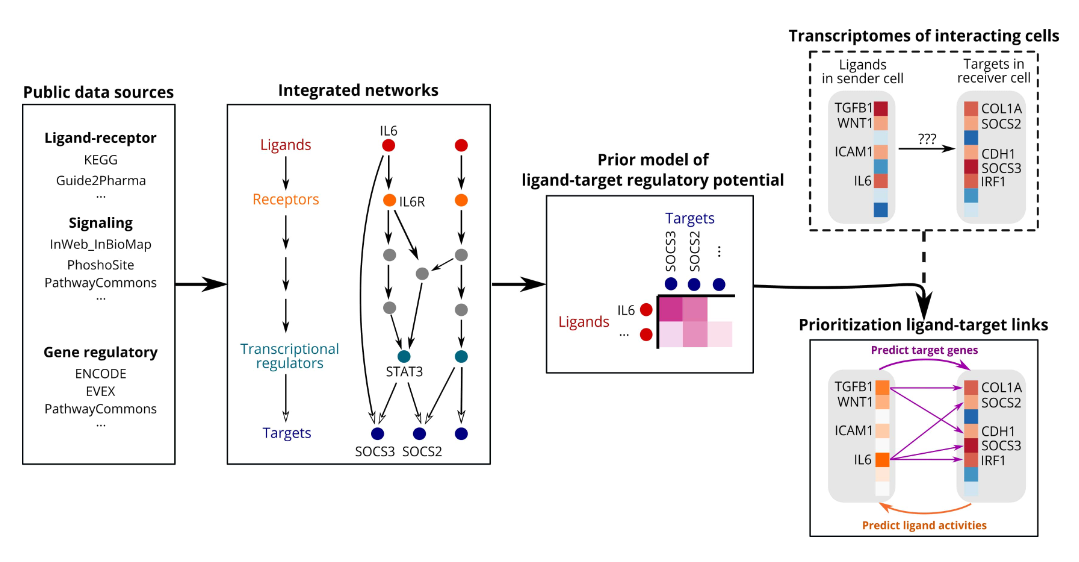
大多数当前研究细胞间通信的计算方法有很大不同,NicheNet 基于配体的基因调控作用,使用的先验知识并结合了细胞内信号传导和转录调控。NicheNet 可以预测哪些配体影响另一个细胞中的表达,哪些靶基因受每个配体的影响以及可能涉及哪些信号传导介质。
R包链接:https://github.com/saeyslab/nichenetr
在所有主要的操作系统上,安装多个R解释器并在它们之间切换是一项很累人的任务,这个工具就是方便解决这个事情的。
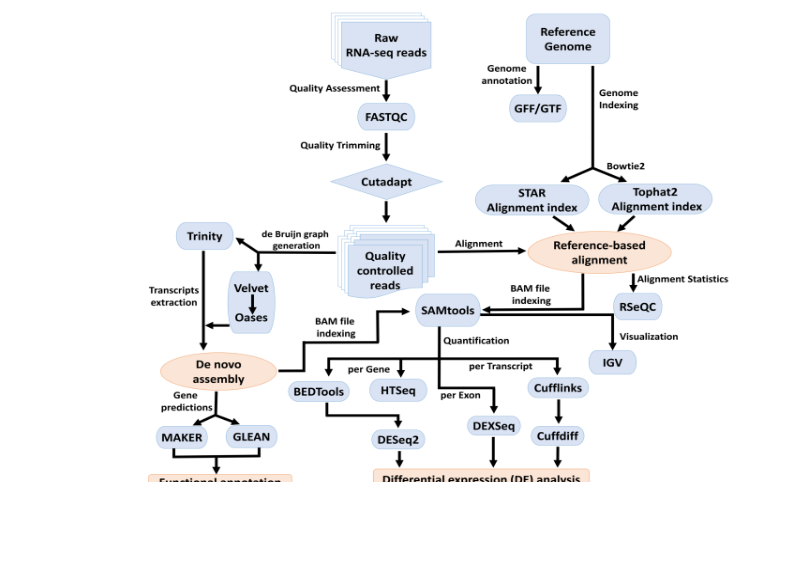
15、BookChapter-RNA-Seq-Analyses - RNA测序数据的设计与分析
本章内容针对RNASeq数据分析提供了全面的概述,包括质量评估,reads的预处理、reads比对,基因定量、可变剪切和差异分析等内容,另外本书中也提供了带有简要描述的代码和数据格式,该工作流的具体使用步骤,详见Github文档。
Github:https://github.com/grimmlab/BookChapter-RNA-Seq-Analyses
Book:https://doi.org/10.1007/978-3-030-62490-3_11
Amazon link: https://www.amazon.com/Generation-Sequencing-Analysis-Materials-Biosciences/dp/3030624897
作者在经过数次测试之后,按照自己的工作经验与偏好,编写出一套成熟的Window开发环境用于开发,并介绍了具体的安装和配置方法。
「Openbiox 生信周刊」运维小队:
@ShixiangWang@kkjtmac@NiEntropy@He-Kai-fly@JnanZhang@Tomcxf@wangdepin
这个周刊每周日发布,同步更新在微信公众号「优雅R」(elegant-r)上。
微信搜索“优雅R”或者扫描二维码,即可订阅。
(完)